OCRツール、Apache Spark、およびその他のApacheHadoopコンポーネントを使用してPDF画像を大規模に処理する方法を学びます。
光学式文字認識(OCR)テクノロジーは、過去20年間で大幅に進歩しました。ただし、その間、OCRをApache Hadoopなどの分散アーキテクチャと組み合わせて、ほぼリアルタイムで多数の画像を処理する取り組みはほとんど、またはまったくありませんでした。
この投稿では、標準のオープンソースツールとApache Spark、Apache Solr、Apache HBaseなどのHadoopコンポーネントを使用して、医療機器情報のユースケースでそれを行う方法を学習します。具体的には、公開データセットを使用して、物語のテキストを検索可能なフィールドに変換します。
この例は医療機器情報に焦点を当てていますが、画像の処理と永続化が必要な他の多くのシナリオに適用できます。たとえば、保険会社は、請求ファイル内のスキャンされたすべてのドキュメントを検索可能にして、請求の解決を向上させることができます。同様に、製造施設のサプライチェーン部門は、部品サプライヤーからのすべての技術データシートをスキャンして、アナリストが検索できるようにすることができます。
ユースケース:医療機器の登録
近年、電子医薬品登録の分野で大きな変化が見られます。 IDMP(医療製品の識別)ISO規格は、製品とその中に含まれる物質を登録するためのそのようなメッセージ形式の1つであり、医薬品ID、パッケージID、およびバッチIDは、不利な経験、違法な場合に製品を追跡するために使用されます輸入、偽造、およびその他のファーマコビジランスの問題。この規格では、新製品を登録する必要があるだけでなく、一般に公開される可能性のあるすべての製品の古い/アーカイブされたファイリングも電子形式で提供する必要があります。
さまざまな企業のIDMP標準に準拠するには、企業はRDBMSや、場合によってはレガシー製品のデータシートなどの複数のデータソースからデータを取得して処理できる必要があります。 Apache Sqoopなどのテクノロジーを介してRDBMSからデータを取り込む方法はよく知られていますが、レガシードキュメント処理にはもう少し作業が必要です。ほとんどの場合、ドキュメントを取り込む必要があり、関連するテキストは、既存のOCRテクノロジーを使用してプログラムで大規模に抽出する必要があります。
データセット
1976年以降に医療機器メーカーから提出されたすべての510(k)ファイリングを含むFDAのデータセットを使用します。食品医薬品化粧品法のセクション510(k)では、登録が必要なデバイスメーカーに通知する必要があります。 FDAは、少なくとも90日前に医療機器を販売する意向を示しています。
この場合、このデータセットはいくつかの理由で役立ちます。
- データは無料でパブリックドメインです。
- データは、2016年7月に施行されるヨーロッパの規制にぴったり適合しています(メーカーは新しいデータ標準に準拠する必要があります)。 FDAの充填物には、IDMPの完全なビューを導き出すことに関連する重要な情報があります。
- ドキュメントの形式(PDF)を使用すると、複数の形式のドキュメントを処理するときに、シンプルでありながら効果的なOCR技術を示すことができます。
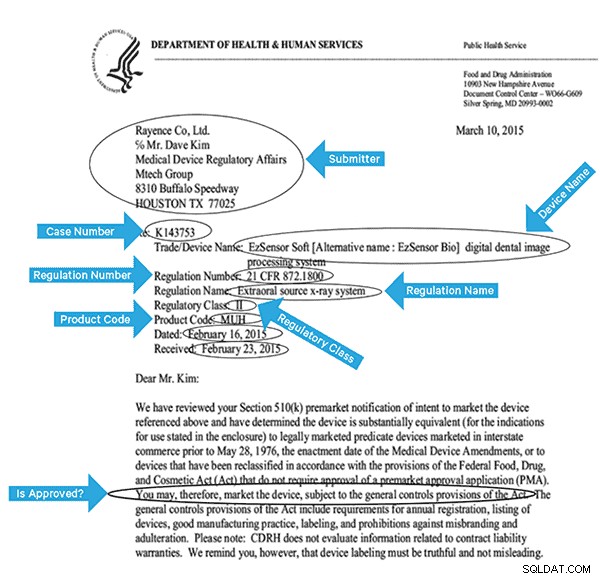
このデータに効果的にインデックスを付けるには、画像からいくつかのフィールドを抽出する必要があります。以下は、抽出できる可能性のあるフィールドを含むサンプルドキュメントです。
高レベルのアーキテクチャ
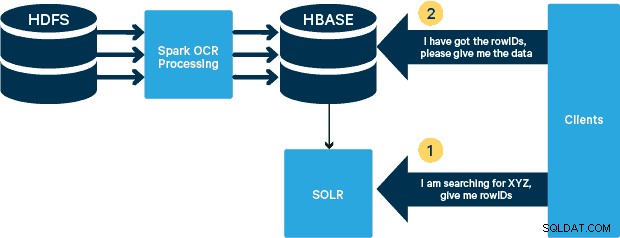
このユースケースでは、PDFはHDFSに保存され、SparkおよびOCRライブラリを使用して処理されます。 (取り込み手順はこの投稿の範囲外ですが、hdfs -dfs -put を実行するのと同じくらい簡単な場合があります またはwebhdfsインターフェースを使用します。)Sparkを使用すると、Spark Streamingアプリケーションでほぼ同じコードを使用してほぼリアルタイムのストリーミングを行うことができます。HBaseは、低遅延のランダムアクセスに最適なストレージメディアであり、画像の保存に最適です。起動する新しいMOB機能。 Cloudera Search(Apache Solr上に構築されている)は、HBaseとネイティブに統合され、セカンダリインデックスを構築できる唯一の検索ソリューションです。
HBaseでの医療機器テーブルの設定
ユースケースのスキーマは単純なままにしておきます。 rowIDはファイル名になり、「info」と「obj」の2つの列ファミリーがあります。 「info」列ファミリーには、画像から抽出したすべてのフィールドが含まれます。 「obj」列ファミリーは、実際のバイナリオブジェクト(この場合はPDF)のバイトを保持します。この場合のテーブルの名前は「mdds」になります。
HBASE-11339で導入されたHBaseMOB(ミディアムオブジェクト)機能を利用します。 MOBを処理するようにHBaseをセットアップするには、いくつかの追加の手順が必要ですが、便利なことに、このリンクに手順があります。
プログラムでHBaseにテーブルを作成する方法はたくさんあります(Java API、REST API、または同様の方法)。ここでは、HBaseシェルを使用して「mdds」テーブルを作成します(わかりやすくするために、意図的に説明的な列ファミリー名を使用します)。 「info」列ファミリーをSolrに複製したいが、MOBデータは複製したくない。
以下のコマンドは、テーブルを作成し、「info」と呼ばれる列ファミリーでレプリケーションを有効にします。オプションREPLICATION_SCOPE => '1'を指定することが重要です そうしないと、HBaseLilyIndexerはHBaseから更新を取得しません。 10MBを超えるオブジェクトには、HBaseのMOBパスを使用します。これを実現するために、MOBの次のパラメータを使用して、「obj」と呼ばれる別の列ファミリも作成します。
IS_MOB => true, MOB_THRESHOLD => 10240000
IS_MOB パラメータは、この列ファミリがMOBを格納できるかどうかを指定し、MOB_THRESHOLD オブジェクトがMOBと見なされるために必要なオブジェクトの大きさを指定します。それでは、テーブルを作成しましょう:
create 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
テーブルが正しく作成されたことを確認するには、HBaseシェルで次のコマンドを実行します。
hbase(main):001:0> describe 'mdds'
Table mdds is ENABLED
mdds
COLUMN FAMILIES DESCRIPTION
{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1', VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
2 row(s) in 0.3440 seconds
スキャンした画像をTesseractで処理する
OCRは、フォントのバリエーション、画像のノイズ、および配置の問題に対処するという点で長い道のりを歩んできました。ここでは、元々HP研究所でプロプライエタリソフトウェアとして開発されたオープンソースのOCRエンジンTesseractを使用します。 Tesseractの開発は、オープンソースソフトウェアとしてリリースされ、2006年からGoogleが後援しています。
Tesseractは、移植性の高いソフトウェアライブラリです。 Leptonica画像処理ライブラリを使用して、灰色または色付きの画像に対して適応しきい値処理を実行することにより、バイナリ画像を生成します。
処理は、従来の段階的なパイプラインに従います。手順の大まかな流れは次のとおりです。
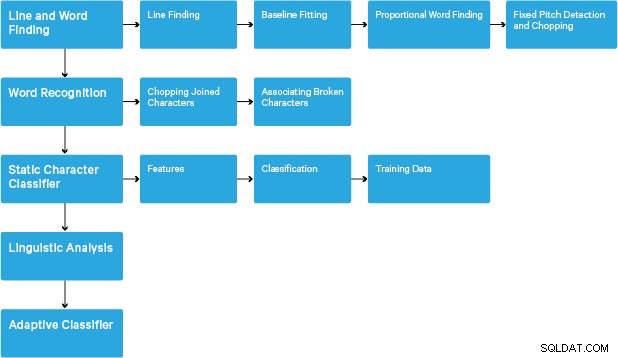
処理は連結成分分析から始まり、見つかった成分が保存されます。この手順は、アウトラインのネスト、および子と孫のアウトラインの数を調べるのに役立ちます。
この段階で、アウトラインは、純粋にネストによって、バイナリラージオブジェクト(BLOB)にまとめられます。 BLOBはテキスト行に編成され、行と領域は固定ピッチまたは比例テキストについて分析されます。テキスト行は、文字間隔の種類に応じて異なる方法で単語に分割されます。固定ピッチのテキストは、文字セルによってすぐに切り刻まれます。プロポーショナルテキストは、明確なスペースとあいまいなスペースを使用して単語に分割されます。
その後、認識は2パスプロセスとして進行します。最初のパスでは、各単語を順番に認識しようとします。満足のいく各単語は、トレーニングデータとして適応分類器に渡されます。次に、適応分類器は、ページの下部にあるテキストをより正確に認識する機会を得ます。適応分類器は、ページの上部近くで貢献するには遅すぎる有用な何かを学習した可能性があるため、2回目のパスがページ上で実行され、十分に認識されなかった単語が再度認識されます。最終段階では、あいまいなスペースを解決し、エックスハイトの対立仮説をチェックして、小文字のテキストを見つけます。
現在の形式のTesseractは完全にユニコード対応であり、いくつかの言語用にトレーニングされています。私たちの調査に基づくと、これはOCRで利用できる最も正確なオープンソースライブラリの1つです。前述のように、TesseractはLeptonicaを使用します。また、Ghostscriptを使用してPDFファイルを画像に分割します。 (選択した画像圧縮形式に分割できます。PNGを選択しました。)これら3つのライブラリはC ++で記述されており、Java / Scalaプログラムから呼び出すには、対応するJavaNativeInterfaceの実装を使用する必要があります。この作業では、JavaPresetsのJNIバインディングを使用します。 (ビルド手順は以下にあります。)Scalaを使用してSparkドライバーを作成しました。
val renderer :SimpleRenderer = new SimpleRenderer( ) renderer.setResolution( 300 ) val images:List[Image] = renderer.render( document )
レプトニカは前のステップで分割された画像を読み込みます。
ImageIO.write(
x.asInstanceOf[RenderedImage],
"png",
imageByteStream
)
val pix: PIX = pixReadMem (
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ),
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity( )
) 次に、TesseractAPI呼び出しを使用してテキストを抽出します。ここでは、ドキュメントが英語であると想定しているため、Initメソッドの2番目のパラメーターは「eng」です。
val api: TessBaseAPI = new TessBaseAPI( ) api.Init( null, "eng" ) api.SetImage(pix) api.GetUTF8Text().getString()
画像が処理された後、テキストからいくつかのフィールドを抽出し、HBaseに送信します。
def populateHbase (
fileName:String,
lines: String,
pdf:org.apache.spark.input.PortableDataStream) : Unit =
{
/** Configure and open a HBase connection */
val mddsTbl = _conn.getTable( TableName.valueOf( "mdds" ));
val cf = "info"
val put = new Put( Bytes.toBytes( fileName ))
/**
* Extract Fields here using Regexes
* Create Put objects and send to HBase
*/
val aAndCP = """(?s)(?m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe: (\w\d\d\d\d\d\d).*""".r
……..
lines match {
case
aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "submitter_info" ),
Bytes.toBytes( addr ) ).add( Bytes.toBytes( cf ),
Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum ))
case _ => println( "did not match a regex" )
}
…….
lines.split("\n").foreach {
val regNumRegex = """Regulation Number:\s+(.+)""".r
val regNameRegex = """Regulation Name:\s+(.+)""".r
……..
…….
_ match {
case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "reg_num" ),
…….
…..
case _ => print( "" )
}
}
put.add( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines ))
val pdfBytes = pdf.toArray.clone
put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( "pdf" ), pdfBytes )
mddsTbl.put( put )
…….
}
上記のコードをよく見ると、PutオブジェクトをHBaseに送信する直前に、生のPDFバイトをテーブルの「obj」列ファミリーに挿入します。抽出されたフィールドと生の画像のストレージレイヤーとしてHBaseを使用します。これにより、必要に応じて、アプリケーションが元の画像をすばやく簡単に抽出できるようになります。完全なコードはここにあります。 (標準のHBase APIを使用してHBaseのPutオブジェクトを作成しましたが、実際の本番システムでは、SparkRDDからHBaseへのバッチ更新を可能にするSparkOnHBaseAPIの使用を検討するのが賢明です。)
>実行パイプライン
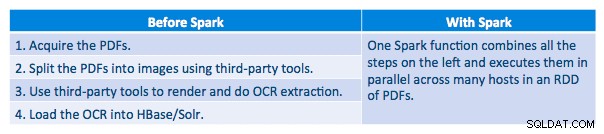
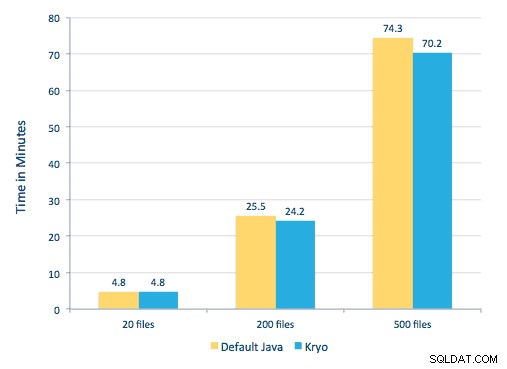
各PDFをシリアルフレームワークで処理することができました。処理を拡張するために、Sparkを使用してこれらのPDFを分散して処理することを選択しました。次のグラフは、この処理のさまざまな段階を組み合わせて、ワークフローをSparkからの単純なマクロ呼び出しに変換し、データをHBaseにロードする方法を示しています。
また、シリアル化方法を比較しようとしましたが、データセットでは、パフォーマンスに大きな違いは見られませんでした。
環境設定
使用したハードウェア:15 GBのメモリ、4つのvCPU、2x40GBのSSDを備えた5ノードクラスター
処理にC++ライブラリを使用していたため、ここにあるJNIバインディングを使用しました。
javaCPPプリセットからTesseractとLeptonicaのJNIバインディングを構築します:
-
- すべてのノード:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel -
git clone https://github.com/bytedeco/javacpp-presets.git -
cd javacpp-presets - ビルドLeptonica。
cd leptonica ./cppbuild.sh install leptonica cd cppbuild/linux-x86_64/leptonica-1.72/ LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && sudo make install cd ../../../ mvn clean install cd ..
- Tesseractを構築します。
- すべてのノード:
cd tesseract ./cppbuild.sh install tesseract cd tesseract/cppbuild/linux-x86_64/tesseract-3.03 LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && make install cd ../../../ mvn clean install cd ..
- javaCPPプリセットをビルドします。
mvn clean install --projects leptonica,tesseract
Ghostscriptを使用してPDFから画像を抽出します。ここで使用されているTesseractとLeptonicaのバージョンに対応するGhostscriptをビルドする手順は次のとおりです。 (Ghostscriptがパッケージマネージャーを介してシステムにインストールされていないことを確認してください。)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gz tar zxvf ghostscript-9.16.tar.gz cd ghostscript-9.16 ./autogen.sh && ./configure --prefix=/usr --disable-compile-inits --enable-dynamic sudo make && make soinstall && install -v -m644 base/*.h /usr/include/ghostscript && ln -v -s ghostscript /usr/include/ps (Depending on your ldpath setting, you may have to do) : sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
必要なすべてのライブラリがクラスパスにあることを確認してください。関連するすべてのjarファイルをlibというディレクトリに配置します。カンマは以下で重要です:
$ for i in `ls lib/*`; do export MY_JARS=./$i,$MY_JARS; done tesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
次のようにSparkプログラムを呼び出します。ネイティブGhostscriptライブラリのextraLibraryPathを指定する必要があります。 Tesseractにはもう1つのconfが必要です。
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor- cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --conf spark.executorEnv.TESSDATA_PREFIX=/home/vsingh/javacpp- presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --conf spark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/ lib/htrace-core-3.1.0-incubating.jar --driver-class-path /etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0- incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer --conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Solrコレクションの作成
Solrは、LilyHBaseIndexerを介してHBaseと非常にシームレスに統合されます。 Lily IndexerとHBaseの統合がどのように行われるかを理解するには、「HBaseレプリケーションとLilyHBaseインデクサーについて」セクションの以前の投稿でブラッシュアップできます。
以下に、インデックスを作成するために実行する必要のある手順の概要を示します。
- サンプルschema.xml構成ファイルを生成します:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg -
$HOME/solrcfgのschema.xmlファイルを編集します 、コレクションに必要なフィールドを指定します。完全なファイルはここにあります。 - Solr構成をZooKeeperにアップロードします:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - 2つのシャード(-s 2)と2つのレプリカ(-r 2)を使用してSolrコレクションを生成します。
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
上記のコマンドでは、2つのシャード(-s 2)と2つのレプリカ(-r 2)パラメーターを使用してSolrコレクションを作成しました。コーパスにはパラメータで十分でしたが、実際の展開では、ここでの説明の範囲外の他の考慮事項に基づいて数を設定する必要があります。
インデクサーの登録
この手順は、インデクサーとHBaseレプリケーションを追加および構成するために必要です。以下のコマンドは、ZooKeeperを更新し、mdds_indexerをHBaseのレプリケーションピアとして追加します。また、構成をZooKeeperに挿入します。これは、LilyHBaseIndexerがSolrの適切なコレクションを指すために使用します。 |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
引数:
-
-n mdds_indexer–ZooKeeperに登録されるインデクサーの名前を指定します -
-c indexer-config.xml–インデクサーの動作を指定する構成ファイル -
-cp solr.zk=localhost:2181/solr–ZooKeeperおよびSolr構成の場所を指定します。これは、ZooKeeperの環境固有の場所で更新する必要があります。 -
-cp solr.collection=mdds_collection–更新するコレクションを指定します。 collection1を作成したSolr構成ステップを思い出してください。
index-config.xml この場合、ファイルは比較的簡単です。調べるテーブルをインデクサーに指定するだけで、マッパーとして使用されるクラス(com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper )、およびMorphline構成ファイルの場所。デフォルトでは、mapping-typeは rowに設定されています 、この場合、Solrドキュメントは完全な行になります。 Param name="morphlineFile" Morphlines構成ファイルの場所を指定します。場所はMorphlinesファイルの絶対パスである可能性がありますが、Cloudera Managerを使用しているため、相対パスをmorphlines.confとして指定します。
hbase-indexer構成ファイルの内容はここにあります。
LilyHBaseインデクサーの構成と起動
Lily HBaseインデクサーを有効にする場合、このインデクサーが医療機器テーブルの更新を解析し、関連するすべてのフィールドを抽出できるようにするMorphlines変換ロジックを指定する必要があります。 [サービス]に移動し、前に追加したLilyHBaseIndexerを選択します。 [構成]->[表示と編集]->[サービス全体]->[モーフライン]を選択します 。 Morphlinesファイルをコピーして貼り付けます。
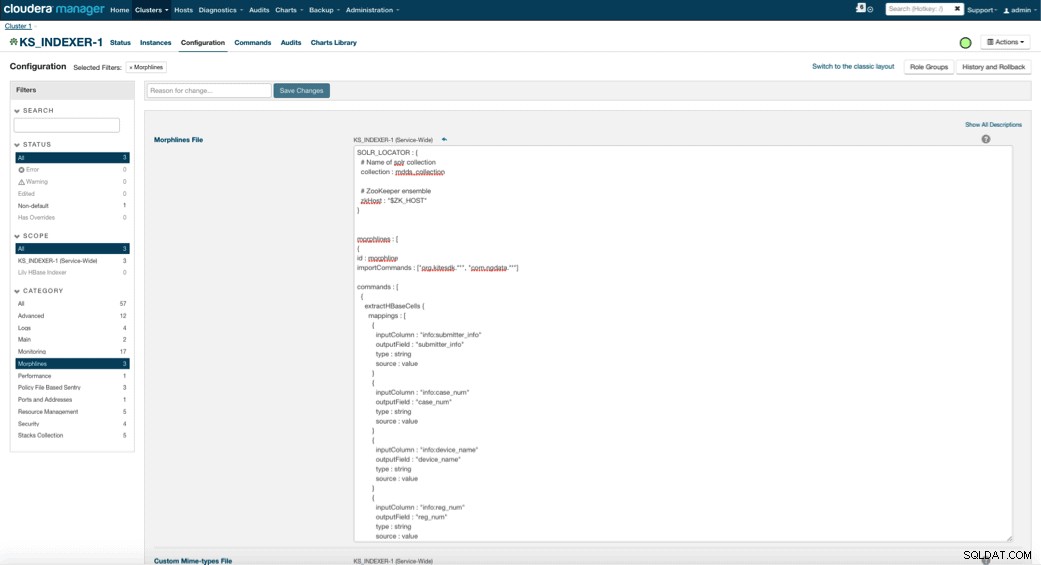
医療機器モーフラインライブラリは、次のアクションを実行します。
-
extractHBaseCellsを使用してHBaseメールイベントを読み取ります コマンド -
convertTimestampを使用して、日付/タイムスタンプをSolrが理解できるフィールドに変換します コマンド - schema.xmlで指定しなかった余分なフィールドをすべて、
sanitizeUknownSolrFieldsで削除します。 コマンド
このMorphlinesファイルのコピーをここからダウンロードしてください。
重要な注意点の1つは、idフィールドがLilyHBaseIndexerによって自動的に生成されることです。この設定は、unique-key-field属性を指定することにより、上記のindex-config.xmlファイルで構成できます。 idのデフォルト名をそのままにしておくことをお勧めします。上記のxmlファイルでは指定されていないため、デフォルトのidフィールドが生成され、RowIDの組み合わせになります。
データへのアクセス
インデックス付きの画像にアクセスするには、多くのビジュアルツールを選択できます。 HUEとSolrGUIはどちらも非常に優れたオプションです。 HBaseは、GUIからだけでなく、HBaseシェル、API、さらには単純なスクリプト手法を介した、さまざまなアクセス手法も可能にします。
Solrとの統合により、優れた柔軟性が得られ、データの非常に単純な検索オプションと高度な検索オプションを提供することもできます。たとえば、電子メールオブジェクト内のすべてのフィールドがSolrに格納されるように、Solr schema.xmlファイルを構成すると、ユーザーは、ストレージスペースと計算の複雑さを犠牲にして、単純な検索で完全なメッセージ本文にアクセスできます。または、IDなどの限られた数のフィールドのみを保管するようにSolrを構成することもできます。これらの要素を使用すると、ユーザーはSolrをすばやく検索し、rowIDを取得できます。これを使用して、HBase自体から個々のフィールドまたは画像全体を取得できます。
上記の例では、rowIDのみをSolrに格納しますが、画像から抽出されたすべてのフィールドにインデックスを付けます。このシナリオでSolrを検索すると、HBase行IDが取得され、これを使用してHBaseにクエリを実行できます。このタイプのセットアップは、ストレージコストを低く抑え、Solrのインデックス機能を最大限に活用するため、Solrにとって理想的です。
サンプルクエリ
以下は、アプリケーションからSolrに実行できるクエリの例です。クライアントは最初にSolrインデックスを照会し、HBaseからrowIDを返すという考え方です。次に、残りのフィールドや元の生の画像についてHBaseにクエリを実行します。
- 次の日付の間に提出されたすべてのドキュメントを教えてください:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01-06T23:59:59.999Z TO 2010-02-06T23:59:59.999Z]
- モバイルX線システムの規制名で提出されたドキュメントを教えてください:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile x-ray system
- 中国のメーカーから提出されたすべてのドキュメントを教えてください:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
SolrドキュメントのIDは、HBaseの行IDです。クエリの2番目の部分は、データを抽出するためのHBaseへの送信です(必要に応じて生のPDFを含む)。
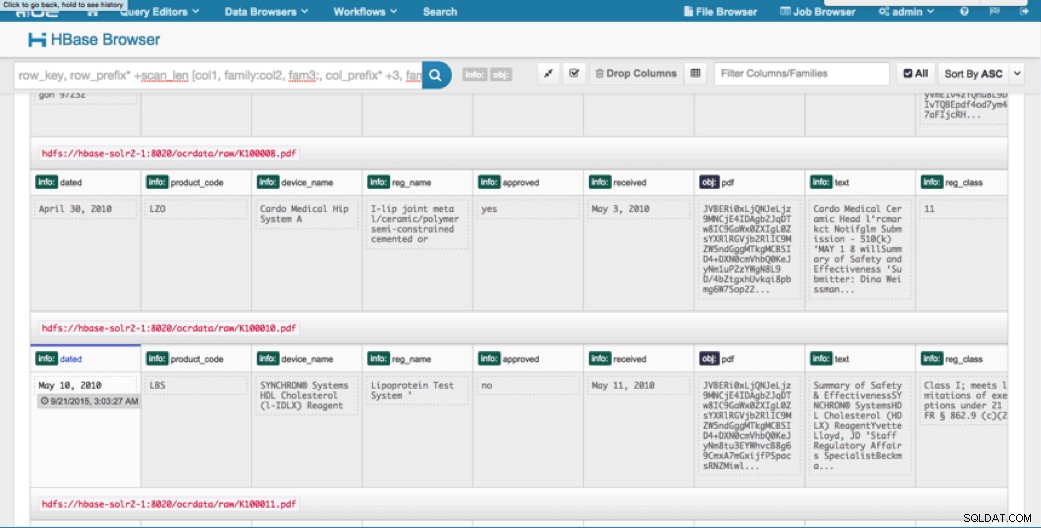
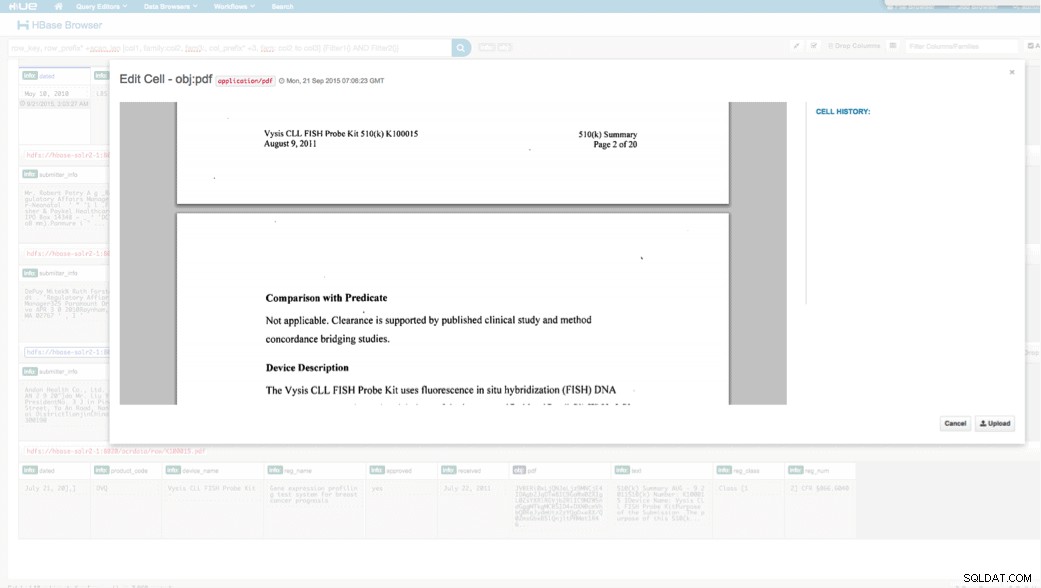
HUE経由のアクセス
アップロードされたデータは、HUEのHBaseブラウザーを介して表示できます。 HUEの優れた点の1つは、PDFのバイナリを検出し、クリックするとレンダリングできることです。
以下は、HBase行の解析済みフィールドのビューのスナップショットと、obj列ファミリーにMOBとして格納されているPDFオブジェクトの1つのレンダリングビューです。
結論
この投稿では、標準のオープンソーステクノロジーを使用して、スケーラブルなSparkプログラムを使用してスキャンされたドキュメントでOCRを実行し、高速検索のためにHBaseに保存し、Solrで抽出された情報にインデックスを付ける方法を示しました。次のことは明らかです:
- メッセージ指定形式を指定すると、フィールドと値のペアを抽出して、Solrを介して検索できるようにすることができます。
- データのこれらのフィールドは、レガシーデータを電子化するというIDMP要件を満たすことができます。これは、来年中に発効します。
- フィールドと生の画像はHBaseに永続化され、標準のAPIを介してアクセスできます。
スキャンしたドキュメントを処理し、データを企業内の他のさまざまなソースと組み合わせる必要がある場合は、Spark、HBase、Solr、およびTesseractとLeptonicaの組み合わせを検討してください。それはあなたにかなりの時間とお金を節約するかもしれません!
Jeff Shmainは、Clouderaのシニアソリューションアーキテクトです。彼は16年以上の金融業界での経験があり、証券取引、リスク、規制について深い理解を持っています。過去数年間、彼は世界最大の投資銀行10行のうち8行でさまざまなユースケースの実装に取り組んできました。
Vartika Singhは、Clouderaのシニアソリューションコンサルタントです。彼女は、応用機械学習とソフトウェア開発で12年以上の経験があります。
